W trakcie prowadzenia badań genetycznych często napotykamy niewystarczające próbki RNA, na przykład do badania niewielkich anatomicznych guzów jamy ustnej, nawet próbki pojedynczych komórek oraz próbki określonych mutacji genów, które są transkrybowane na bardzo niskim poziomie w komórkach ludzkich.Oczywiście w przypadku testu na COVID-19, jeśli wymazy nie znajdą się we właściwym miejscu lub za mało razy podczas pobierania próbek, wielkość próby będzie bardzo mała, dlatego Komisja Zdrowia i Planowania Rodziny wyszła dwa dni temu i zdał test, a jeśli próbnik kwasu nukleinowego nie pobrał sześciu próbek, możesz to zgłosić.
Czułość odczynnika jest ważna, ponieważ mamy ten lub inny problem, więc co możemy zrobić, aby poprawić czułość RT-PCR?
Zanim omówimy możliwe rozwiązania, wspomnijmy o dwóch dużych komplikacjach związanych z sytuacją, o której właśnie wspomnieliśmy.
Przede wszystkim martwimy się utratą RNA, gdy w naszej próbce mamy tylko kilka populacji komórek.W przypadku stosowania tradycyjnych metod rozdzielania i oczyszczania, takich jak metoda kolumnowa lub metoda wytrącania kwasów nukleinowych, istnieje duże prawdopodobieństwo, że kilka próbek zostanie utraconych.Jednym z rozwiązań jest dodanie cząsteczki nośnika, takiej jak tRNA, ale nawet wtedy nie ma gwarancji, że nasz eksperyment odzyskiwania przebiegnie pomyślnie.
Więc jaki jest lepszy sposób?Dobrą opcją dla hodowanych komórek lub próbek mikroanatomicznych jest zastosowanie bezpośredniej lizy.
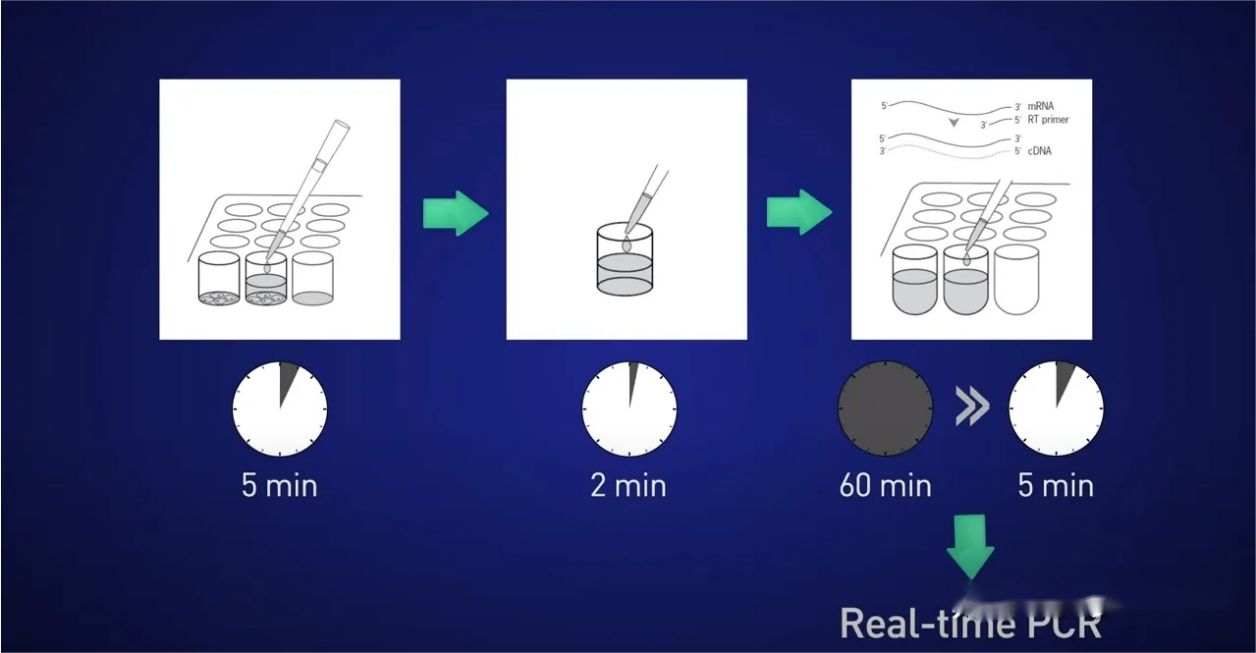
Pomysł polega na podzieleniu komórek na 5 minut, uwolnieniu RNA do roztworu, następnie zatrzymaniu reakcji na 2 minuty, następnie dodaniu lizatu bezpośrednio do reakcji odwrotnej transkrypcji, tak aby żaden RNA nie został utracony, a na koniec bezpośrednie umieszczenie otrzymanego cDNA w reakcję w czasie rzeczywistym.
Ale co, jeśli z powodu ograniczonego punktu początkowego lub niewielkiej ilości docelowej ekspresji genów możemy poddać recyklingowi cały RNA i nadal nie dostarczyć wystarczającej liczby szablonów, aby uzyskać dobry sygnał w czasie rzeczywistym?
W takim przypadku etap wstępnej amplifikacji może być bardzo użyteczny.
Poniżej przedstawiono schemat zwiększania czułości po odwrotnej transkrypcji.Przed rozpoczęciem musimy zapytać, które cele nas interesują, aby zaprojektować specyficzne startery dla tych celów do wstępnej amplifikacji.
Można to osiągnąć, tworząc mieszany starter z maksymalnie 100 parami starterów i cyklem reakcji od 10 do 14 razy.Dlatego do wstępnej amplifikacji otrzymanego cDNA potrzebny jest Master Mix specjalnie zaprojektowany dla tego wymagania.
Powodem ustalenia liczby cykli między 10 a 14 jest to, że ta ograniczona liczba cykli zapewnia losowość między różnymi celami, co ma kluczowe znaczenie dla naukowców potrzebujących ilościowych informacji molekularnych.
Po wstępnej amplifikacji możemy uzyskać dużą ilość cDNA, dzięki czemu czułość wykrywania na końcu jest znacznie poprawiona, a nawet możemy rozcieńczyć próbkę i przeprowadzić wiele reakcji PCR w czasie rzeczywistym, aby wyeliminować ewentualne przypadkowe błędy.
Czas postu: 11 kwietnia 2023 r